1. Tabellen mit R
1.1 Tabelle: Verteilung einer kategorialen Variable
Der Befehl freq() aus dem Package
summarytools erzeugt eine Tabelle, aus der ihr direkt
Anteile, kumulierte Prozente und absolute Werte ablesen könnt. Die
validen Prozente geben die Prozentanteile der jeweiligen Kategorien ohne
Einbezug der NA an. Achtung: Bei einigen Mac Usern
wird für eine erfolgreiche Installation/Aktivierung des
Summarytools-Packages der vorherige Download und Installation des
XQuartz Programms benötigt. Der Downloadlink wird den Mac Usern in der
Fehlermeldung hinterlegt. Alternativ findet man das Programm aber auch
über Folgenden Link:https://www.xquartz.org/
Wir inspizieren zunächst die Variable intmig
(Interesse an Migration und Integration) und stellen ihre
Verteilung tabellarisch dar:
attributes(kursdata_anon$intmig)
## $label
## [1] "interesse: Migration und Integration"
##
## $format.stata
## [1] "%9.0g"
##
## $class
## [1] "haven_labelled" "vctrs_vctr" "double"
##
## $labels
## sehr etwas gar nicht
## 1 2 3
library(summarytools)
freq(kursdata_anon$intmig)
## Frequencies
##
## Freq % Valid % Valid Cum. % Total % Total Cum.
## ----------- ------ --------- -------------- --------- --------------
## 1 31 57.41 57.41 57.41 57.41
## 2 22 40.74 98.15 40.74 98.15
## 3 1 1.85 100.00 1.85 100.00
## <NA> 0 0.00 100.00
## Total 54 100.00 100.00 100.00 100.00
Meistens ist es sinnvoll, kategoriale Variablen vor Verwendung des
freq() Befehls zu faktorisieren, damit die Ausprägungen
in der Tabelle ersichtlich werden (dieser Effekt tritt allerdings nur
dann ein, wenn (a) entsprechende Wertelabel in der Variable
hinterlegt sind - was hier der Fall ist - und (b) die
Faktorisierungsvariante as_factor() (mit Unterstrich)
zur Klassenspezifikation verwendet wird).
kursdata_anon$intmig <- as_factor(kursdata_anon$intmig)
freq(kursdata_anon$intmig)
## Frequencies
##
## Freq % Valid % Valid Cum. % Total % Total Cum.
## --------------- ------ --------- -------------- --------- --------------
## sehr 31 57.41 57.41 57.41 57.41
## etwas 22 40.74 98.15 40.74 98.15
## gar nicht 1 1.85 100.00 1.85 100.00
## <NA> 0 0.00 100.00
## Total 54 100.00 100.00 100.00 100.00
Der freq() Befehl eignet sich für die tabellarische
Darstellung von Variablen mit einer überschaubaren Anzahl Ausprägungen.
Die mit freq() erstellten Tabellen sind jedoch nicht
direkt publikationswürdig und müssen noch weiterverarbeitet werden. Dies
erfolgt am besten in Word. Dazu schreibst du die per
freq() generierte Tabelle in ein neues Objekt und
speicherst dies als “html” File.
tab_intmig<-freq(kursdata_anon$intmig)
print(tab_intmig, method="browser", file="intmig.html")
Ein File namens “intmig.html” ist nun automatisch in der Working
Directory abgespeichert (wenn du keine Working Directory gesetzt hast,
wird das File entweder am Speicherort des Skripts oder einfach unter
“Documents” abgespeichert). Dieses File kannst du nun in Word öffnen und
dort die Tabelle publikationswürdig aufbereiten. Alternativ kann der
Output-File direkt im .doc-Format angelegt werden (was manchmal etwas
besser, machmal etwas schlechter formatierbare Tabellen produziert). Im
print()-Befehl muss dazu das
file()-Argument angepasst werden:
file="intmig.doc"
Bei der Weiterverarbeitung bzw. externen Formatierung in Word ist zu
achten auf:
- Titel
- Ggf. Fragestellung in Untertitel
- Klare Zeilen- und Spaltennamen (Sprache konsistent Deutsch
oder Englisch)
- Quellenangabe der Daten
- Kumulierte Prozente nur bei mindestens ordinalen Variablen
- Ein oder Zwei Nachkommastellen
- Totale Prozente werden in der Regel nicht ausgewiesen
- Die Zeile ‘keine Angabe’ bzw. ‘NA’ wird manchmal weggelassen und
stattdessen in der Note ein Zusatz angefügt: “an …[hier gesamte
Samplegrösse einfügen] fehlende Werte: keine Angabe”
1.2 Tabelle: Verteilung einer metrischen Variablen
Manchmal kann es sinnvoll sein, auch die Verteilung einer metrischen
Variable als Häufigkeitstabelle darzustellen. Dazu müssen wir die
Variable allerdings meistens zuerst klassifizieren. Zwar gibt es
bestimmte Regeln für solche Klassifizierungsgsprozesse (siehe unten),
aber auch viele Freiheiten, die in Abhängigkeit der
Verteilungseigenschaften und des Erkenntnisinteresses ausgestaltet
werden müssen.
Exkurs: Klassifizieren und Kategorisieren
Wir fassen Werte einer metrischen Variable für die Tabellierung
insbesondere dann zusammen, wenn viele Ausprägungen vorliegen. 10er oder
kleinere Skalen dagegen werden häufig auch nicht-klassifiziert
tabellarisch dargestellt. Wir fassen ebenfalls Werte zusammen, wenn wir
Interesse an einem kategorialen Konzept (z.B. “Armut”) haben, dem eine
metrische Skala (z.B. “Einkommen”) zugrunde liegt, oder wenn unser
Interesse, wie im Beispiel unten, explizit auf Tendenzen ausgerichtet
ist.
Beim Klassifizieren steht die Zusammenfassung
von Ausgangswerten im Vordergrund. Sowohl das Konzept als auch das
metrische Niveau bleiben dabei oft (letzteres in den Grenz- oder
Mittelwerten) erhalten. Beispiel: Einkommen -> 0-2000 CHF; 2001-4000
CHF usw.
Bei der Kategorisierung steht die Bildung neuer
Kategorien - oft auch ein neues Konzept - im Vordergrund. Die Metrik
geht dabei fast immer verloren. Beispiel: Einkommen -> “Arm”; “nicht
Arm”. Die Grenzen zwischen Kategorisieren und
Klassifizieren sind fliessend. Oft werden die beiden Begriffe
Synonym verwendet.
Die Klassen/Kategorien sollten geschlossen nach unten und oben,
disjunkt, erschöpfend und möglichst gleich breit sein
Wir klassifizieren nun die Variable Lebenszufriedenheit
(lezufr) und stellen sie anschliessend wiederum mit
freq() tabellarisch dar:
kursdata_anon$lezufr_kls[kursdata_anon$lezufr >= 0 & kursdata_anon$lezufr <= 33] <- "unzufrieden"
kursdata_anon$lezufr_kls[kursdata_anon$lezufr >= 34 & kursdata_anon$lezufr <= 66] <- "zufrieden"
kursdata_anon$lezufr_kls[kursdata_anon$lezufr >= 67] <-"sehr zufrieden"
freq(kursdata_anon$lezufr_kls)
## Frequencies
##
## Freq % Valid % Valid Cum. % Total % Total Cum.
## -------------------- ------ --------- -------------- --------- --------------
## sehr zufrieden 36 67.92 67.92 66.67 66.67
## unzufrieden 3 5.66 73.58 5.56 72.22
## zufrieden 14 26.42 100.00 25.93 98.15
## <NA> 1 1.85 100.00
## Total 54 100.00 100.00 100.00 100.00
Zwei Drittel (67%) der Befragten aus dem Kurs stufen sich als ‘sehr
zufrieden’ ein (mit Werten zwischen 67 und 100). Etwas weniger als ein
Viertel (bzw. 25%) liegen im Bereich einer ‘zufriedenen’
Selbsteinschätzung. 8% der Befragten haben einen Wert zwischen 0 und 33
gewählt und sind daher als ‘unzufrieden’ klassifiziert (oder eher
“kategorisiert”? Ist ein Grenzfall).
Problem: R ordnet die Kategorien einer Variable bei deren Neubildung
nicht entsprechend der Skala der zugrunde liegenden Variable, sondern
alphabetisch. Dadurch weicht die Reihung in der Tabelle nun unschön von
der ordinalen Logik der klassifizierten Variable ab. Der Befehl
factor(), bzw. dessen sub-Befehl
levels=c(), kann hier Abhilfe schaffen: Hiermit können
wir die Reihenfolge der Kategorien definieren.
kursdata_anon$lezufr_kls <- factor(kursdata_anon$lezufr_kls,
levels = c("unzufrieden", "zufrieden", "sehr zufrieden"))
freq(kursdata_anon$lezufr_kls)
## Frequencies
##
## Freq % Valid % Valid Cum. % Total % Total Cum.
## -------------------- ------ --------- -------------- --------- --------------
## unzufrieden 3 5.66 5.66 5.56 5.56
## zufrieden 14 26.42 32.08 25.93 31.48
## sehr zufrieden 36 67.92 100.00 66.67 98.15
## <NA> 1 1.85 100.00
## Total 54 100.00 100.00 100.00 100.00
Auch diese Tabelle speichern wir zur externen Weiterverarbeitung bzw.
Formatierung:
tab_lekl<-freq(kursdata_anon$lezufr_kls)
print(tab_lekl, method="browser", file="lekl.html")
Zusammenfassend die Arbeitsschritte der Häufigkeitstabellen
mit freq():
- Klassifikations- oder Kategorisierungsentscheidung (bei metrischen
Variablen)
- Variable generieren (bei metrischen Variablen)
- Faktorisieren (wenn nötig)
- Ggf. Kategoriennamen festlegen
- Häufigkeitstabelle erstellen (mit freq)
- Häufigkeitstabelle speichern
- Häufigkeitstabelle formatieren (extern)
1.3 Tabellen: Verteilungseigenschaften metrischer Variablen –
«Stichprobenstatistik»
Tabellen sind ebenfalls ein nützliches Vehikel, um die Parameter von
Verteilungen mehrerer (metrischer) Variablen kompakt darzustellen.
Hierfür ist z.B. das stargazer Package geeignet.
Wir erstellen einen Teildatensatz mit den darzustellenden Variablen.
Wir wählen die metrischen Variablen lezufr,
llezufr, leftright und
alter aus dem Kursdatensatz aus.
library(dplyr)
kursdata_T <- select(kursdata_anon, lezufr, llezufr, leftright, alter)
Wir aktivieren stargazer() und führen den Befehl
aus:
library(stargazer)
##
## Please cite as:
## Hlavac, Marek (2022). stargazer: Well-Formatted Regression and Summary Statistics Tables.
## R package version 5.2.3. https://CRAN.R-project.org/package=stargazer
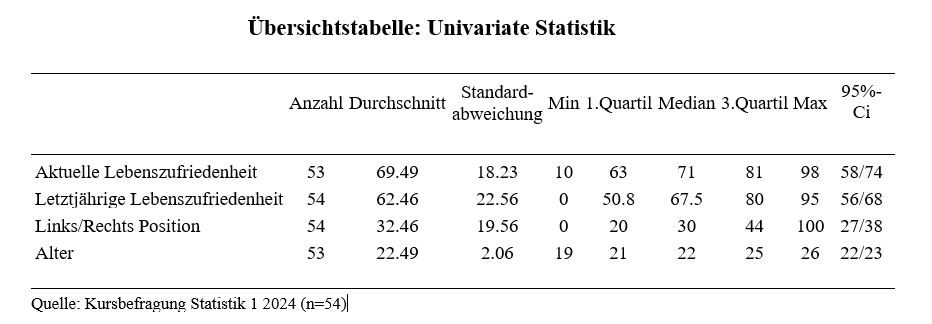
stargazer(as.data.frame(kursdata_T), median = TRUE, iqr = TRUE, type = "text",
title = "Übersichtstabelle: Univariate Statistik", digits = 2)
##
## Übersichtstabelle: Univariate Statistik
## ============================================================
## Statistic N Mean St. Dev. Min Pctl(25) Median Pctl(75) Max
## ------------------------------------------------------------
## lezufr 54 66.37 29.18 -99 61.5 71 80.8 98
## llezufr 54 62.46 22.56 0 50.8 67.5 80 95
## leftright 54 32.46 19.56 0 20 30 44 100
## alter 53 22.49 2.06 19 21 22 25 26
## ------------------------------------------------------------
Mit stargazer() erstellte Tabellen unterscheiden
sich grundlegend von den freq()-Tabellen. Die Variablen
sind bei stargazer() in den Zeilen angesiedelt und die
Parameter der Verteilungen, statt die Verteilungen selbst, werden
kenntlich gemacht. Solche Übersichtstabellen dienen nicht nur der
kompakten Darstellung von Übersichtsstatistiken, sondern können auch bei
der Dateninspektion hilfreich sein: In unserem Beispiel hat sich z.B.
ein unerwünschter, falsch codierter Wert eingeschlichen (-99), den wir
im Rahmen dieser Datenbearbeitung bisher übersehen hatten. Wir
rekodieren ihn getreu der Notation von R als NA:
kursdata_anon$lezufr[kursdata_anon$lezufr == -99] <- NA
summary(kursdata_anon$lezufr)
## Min. 1st Qu. Median Mean 3rd Qu. Max. NA's
## 10.00 63.00 71.00 69.49 81.00 98.00 1
Wir führen nochmals denselben Befehl durch mit der bereinigten
Variablen.
kursdata_T <- select(kursdata_anon, lezufr, llezufr, leftright, alter)
stargazer(as.data.frame(kursdata_T), median = TRUE, iqr = TRUE, type = "text",
title = "Übersichtstabelle: Univariate Statistik", digits = 2)
##
## Übersichtstabelle: Univariate Statistik
## ============================================================
## Statistic N Mean St. Dev. Min Pctl(25) Median Pctl(75) Max
## ------------------------------------------------------------
## lezufr 53 69.49 18.23 10 63 71 81 98
## llezufr 54 62.46 22.56 0 50.8 67.5 80 95
## leftright 54 32.46 19.56 0 20 30 44 100
## alter 53 22.49 2.06 19 21 22 25 26
## ------------------------------------------------------------
Damit wir diese Tabelle aufbereiten können, speichern wir sie als
Word-Dokument im html-Modus ab:
stargazer(as.data.frame(kursdata_T), median = TRUE, type = "html",iqr = TRUE, out = "stargazer.doc",
title = "Übersichtstabelle: Univariate Statistik", digits = 2)
Ein File namens “stargazer.doc” ist jetzt in eurer Working
Directory abgespeichert. Dieses File könnt ihr mit der
Textverarbeitung öffnen und weiterbearbeiten. Neben der Formatierung
könnten dort auch weitere statistische Parameter manuell zugefügt
werden, wie die 95%-Konfidenzintervalle der Mittelwerte:
ci_lezufr <- t.test(kursdata_anon$lezufr)
ci_lezufr$conf.int
ci_llezufr <- t.test(kursdata_anon$llezufr)
ci_llezufr$conf.int
ci_leftright <- t.test(kursdata_anon$leftright)
ci_leftright$conf.int
ci_alter <- t.test(kursdata_anon$alter)
ci_alter$conf.int
Es gelten die gleichen Formatierungsstandards wie bei der
Häufigkeitstabelle oben, letztlich sollte Eure Stichprobenstatistik bzw.
Übersichtstabelle in etwa so aussehen:
2. Grafische Darstellung
2.0 Einführung
library(ggplot2)
Wir erstellen Grafiken in R/ RStudio mit dem Package
ggplot2. Dies ist ein weit verbreitetes Package mit
einer grossen Auswahl an Gestaltungsmöglichkeiten. Startpunkt ist also
die Installation und Aktivierung von ggplot2.
Alternativ kann man auch das tidyverse Meta-Package
aktivieren, da ggplot2 Teil davon ist. Hinweis: Die
folgenden Befehlssequenzen dienen in erster Linie der didaktischen
Motivation. Verwendet wird in der Praxis schliesslich immer nur die
vollständige Plot-Funktion.
2.1 Barplot
Wir werden uns schrittweise an die Befehlslogik von Darstellungen
anhand von ggplot2 heran tasten. Zuerst widmen wir uns
der Darstellung kategorialer Variablen. An erster Stelle wird eine leere
Grafikvorlage erstellt und der Datensatz bestimmt:
plot_1 <- ggplot(kursdata_anon)
plot_1

Mit aes() werden die relevanten Variablen
aufgerufen. Da wir einen univariaten Plot erstellen, müssen wir nur eine
Variable festlegen (hier:konsum). Die Kategorien der
Variable sollen horizontal angeordnet sein, also die x-Achse
definieren.
kursdata_anon$konsum<-as_factor(kursdata_anon$konsum)
plot_1 <- ggplot(kursdata_anon, aes(x = konsum))
plot_1

Hiermit ist die univariate Befehlsbasis bereits vollständig. Darauf
folgen Zusätze, welche die Art, Beschriftung und Ästhetik der
Darstellung ausmachen. Jeder Zusatz wird mit einem + an
den Basisbefehl angehängt.
Mit den “geoms” wird die Art des Plots bestimmt. In diesem Fall steht
das geom_bar für ein Säulendiagramm (= Barplot). Das
ggplot2-Cheatsheet gibt einen Überblick zu den
verschiedenen geom-Typen bzw. Abbildungsarten.
plot_1 <- ggplot(kursdata_anon, aes(x = konsum))+
geom_bar()
plot_1

Innerhalb des jeweiligen geom() Zusatzes lassen sich
weitere Elemente der Grafik spezifizieren, wie beispielsweise die Farbe,
Breite oder Anzahl der Balken. Auch diesbezüglich findet ihr
geom spezifische Informationen im Cheatsheet. Mit
color bestimmen wird z.B. die Rahmenfarbe, mit fill
die Füllfarbe der Balken.
plot_1 <- ggplot(kursdata_anon, aes(x = konsum))+
geom_bar(colour = "blue", fill = "orange")
plot_1

Innerhalb von labs() können die Achsentitel und der
Plottitel festgelegt werden, mit caption können zusätzliche
Informationen unterhalb des Plots ergänzt werden.
plot_1 <- ggplot(kursdata_anon, aes(x = konsum))+
geom_bar(colour = "blue", fill = "orange")+
labs(x = "Supermarkt", y = "Anzahl", title = "Konsumpräferenz im Kurs",
subtitle = "'Welchen Supermarkt bevorzugen Sie?'",
caption ="Quelle: Kursbefragung Statistik I (n=...)")
plot_1

Mit theme kann der Hintergrund festgelegt werden:
classic() steht in diesem Fall für einen
transparenten/keinen Hintergrund.
plot_1 <- ggplot(kursdata_anon, aes(x = konsum))+
geom_bar(colour = "blue", fill = "orange")+
labs(x = "Supermarkt", y = "Anzahl", title = "Konsumpräferenz im Kurs",
subtitle = "'Welchen Supermarkt bevorzugen Sie?'",
caption ="Quelle: Kursbefragung Statistik I (n=...)")+
theme_classic()
plot_1

Im Barplot ist die Kategorie ‘keine Angabe’ enthalten. Diese
Kategorie schliessen wir nun aus der Darstellung aus. Dazu müssen wir
die Personen, die ‘keine Angabe’ gemacht haben, vorab ausfiltern und
einen neuen, entsprechend reduzierten Datensatz erstellen. Wichtig:
Fallzahl in der Caption anpassen!
library(dplyr)
kursdata_anon_N <- filter(kursdata_anon, konsum != "NA")
plot_1 <- ggplot(kursdata_anon_N, aes(x = konsum))+
geom_bar(colour = "blue", fill = "orange")+
labs(x = "Supermarkt", y = "Anzahl", title = "Konsumpräferenz im Kurs",
subtitle = "'Welchen Supermarkt bevorzugen Sie?'",
caption ="Quelle: Kursbefragung Statistik I (n=...)")+
theme_classic()
plot_1

Nun wollen wir die Kategorien auf der x-Achse umbenennen; die Namen
der beiden Ketten sollen gross geschrieben werden.
plot_1 <- ggplot(kursdata_anon_N, aes(x = konsum))+
geom_bar(colour = "blue", fill = "orange")+
scale_x_discrete(labels = c("Coop", "Migros"))+
labs(x = "Supermarkt", y = "Anzahl", title = "Konsumpräferenz im Kurs",
subtitle = "'Welchen Supermarkt bevorzugen Sie?'",
caption ="Quelle: Kursbefragung Statistik I (n=...)")+
theme_classic()
plot_1

In einem letzten Schritt wollen wir die Häufigkeiten auf der y-Achse
als Anteile ausgeben. Mit aes(y = after_stat(count /
sum(count))) werden die Häufigkeiten auf der y-Achse in
Anteilswerte (0, 0.2, 0,4 etc.) umgewandelt. Innerhalb der
Achsenspezifikation scale_y_continuous() kann dann die
Ausgabe der Anteile über labels = scales::percent in
Prozentwerten angefordert werden. Wir erhalten damit den fertigen
Barplot.
plot_1 <- ggplot(kursdata_anon_N, aes(x = konsum))+
geom_bar(colour = "blue", fill = "orange")+
scale_x_discrete(labels = c("Coop", "Migros"))+
aes(y = after_stat(count / sum(count)))+
scale_y_continuous(labels = scales::percent)+
labs(x = "Supermarkt", y = "Anteil", title = "Konsumpräferenz im Kurs",
subtitle = "'Welchen Supermarkt bevorzugen Sie?'",
caption ="Quelle: Kursbefragung Statistik I (n=...)")+
theme_classic()
plot_1
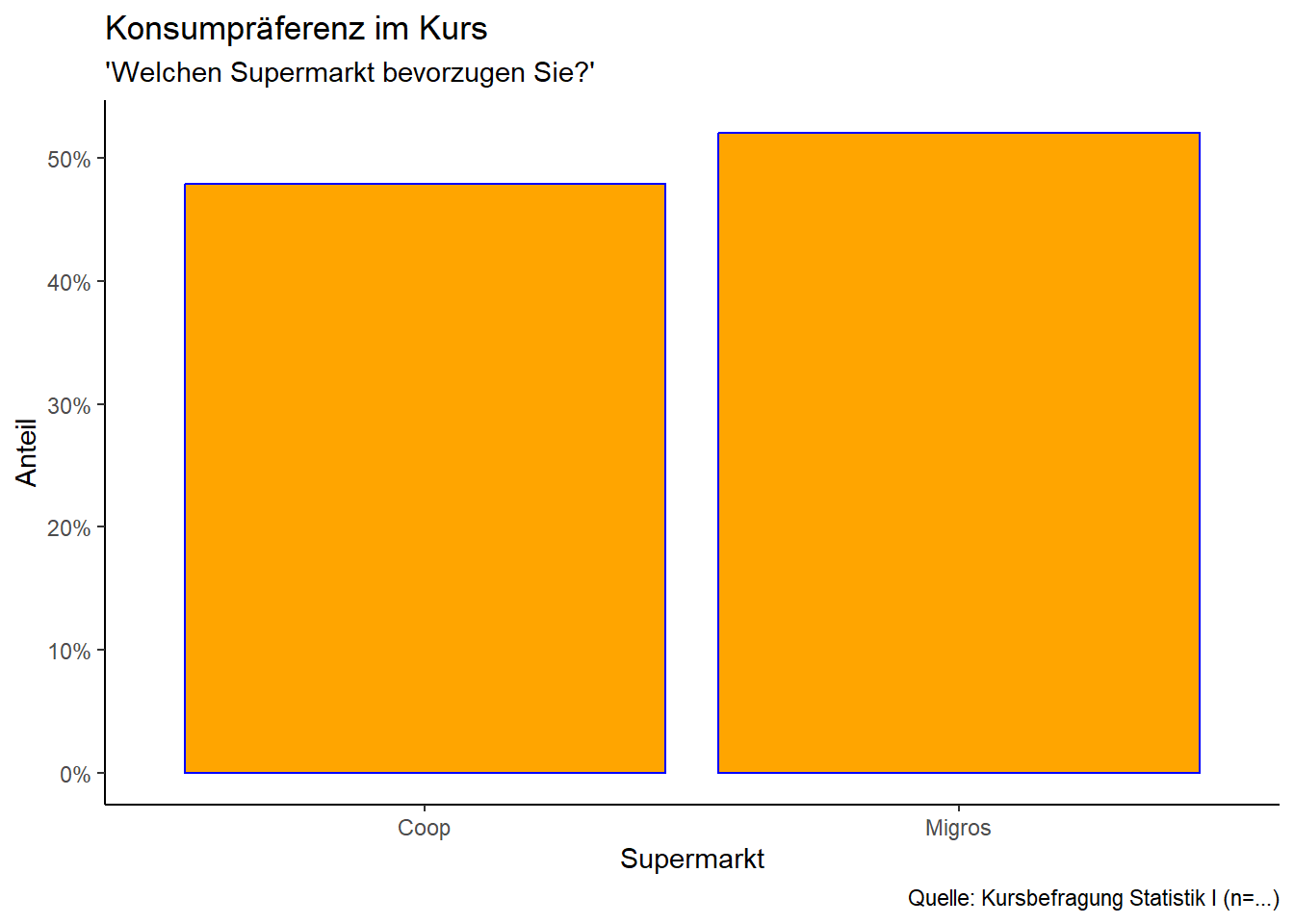
2.2 Histogramm
Histogramme sehen Barplots ähnlich, eignen sich jedoch für die
Visualisierung von metrischen und kontinuierlichen Variablen mit vielen
Ausprägungen. Wir erstellen ein Histogramm mithilfe eines neuen
geom: geom_histogram() für die
metrische Variable lezufr.
In einem ersten Schritt bereinigen wir die Variablen.
kursdata_anon$lezufr[kursdata_anon$lezufr == -99] <- NA
Der Befehl zur Erstellung eines Histogramms ist ähnlich dem
Barplot-Befehl aufgebaut:
plot_2 <- ggplot(kursdata_anon, aes(x = lezufr))+
geom_histogram (fill = "magenta", color="chocolate4", breaks = c(0,10,20,30,40,50,60,70,80,90,100))+
labs(x = "Lebenszufriedenheit",y ="Anzahl",
title = "Lebenszufriedenheit in der Kursbefragung",
subtitle = "Wie zufrieden bist du mit deinem Leben auf einer Skala von 1 (gar nicht) bis 100 (sehr)?",
caption = "Quelle: Kursbefragung Statistik I (n=...)")+
scale_x_continuous(breaks = seq(0,100,10))+
scale_y_continuous(breaks = seq(0,25,5))+
theme_classic()
plot_2

Einige Aspekte müssen jedoch verändert werden.
aes(x = lezufr): Wir erzeugen weiterhin einen
univariaten Plot und müssen deshalb nur eine Variable bestimmen. Wir
nehmen nun aber eine metrische Variable, die Lebenszufriedenheit der
Kursteilnehmenden.
geom_histogram(): Da jetzt ein Histogramm
erzeugt werden soll, müssen wir den Befehl für die Art des Plots
ändern.
breaks =: Hier kann die Anzahl und die Breite der Säulen
und ihre Grenzen bestimmt werden. In diesem Beispiel schliesst die erste
Säule Werte von (0)-(9) ein, die zweite von (10)-(19) etc. Mit
c() werden die einzelnen Grenzen manuell bestimmt.
Alternativ könnten mit seq(0,100,10)
automatisiert 10er Schritte von (0) bis (100) befohlen werden.
bins =: Anstatt breaks. Hier kann die Anzahl
der Säulen bestimmt werden. bins = 20 würde 20 Säulen
anzeigen.
binwidth =: Anstatt bins. Hier kann die Breite
der Säulen festgelegt werden. binwidth = 5 würde eine
Säulenbreite von 5 heissen. Das heisst die erste Säule wäre von (0)-(5),
die zweite von (5)-(10) etc.
scale_x_continuous(): Mit diesem Argument kann
das Skalenniveau für die x-Achse bestimmt werden. In diesem Fall von 0
bis 100 in 10er Schritten.
scale_y_continuous(): Mit diesem Argument kann
das Skalenniveau für die y-Achse bestimmt werden. In diesem Fall von 0
bis 20 in 1er Schritten.
breaks = seq(): Mit diesem Argument können wir
das untere und das obere Limit für die beiden Achsen festlegen.
Ausserdem können wir den Abstand auf den Achsen zwischen zwei
Punktbeschriftungen festlegen.
2.3 Boxplot
Boxplots stellen ebenfalls eine Möglichkeit dar, um Verteilungen
metrischer Variablen zu visualisieren. Allerdings werden hierbei nicht
die Verteilungen selbst, sondern deren Parameter grafisch dargestellt:
Jede der drei Linien des Boxplots steht für ein Quartil der Fälle, also
Q1=25%, Q2=50% und Q3=75%. Der Median, bzw. Q2, wird als dicke Linie in
der Mitte der Box hervorgehoben. Dementsprechend umfasst die Höhe der
Box den Interquartilsabstand “IQR”. Wir gewöhnen uns an, zusätzlich per
Raute den Mittelwert der Verteilung kenntlich zu machen.
Der folgende Boxplot soll die Verteilung der Lebenszufriedenheit im
Kurs zeigen.

Wir erarbeiten den Befehl zur Erstellung des Plots wieder
schrittweise:
Wir erstellen eine leere Grafik und reichern sie mit der metrischen
Variablen lezufr auf der y-Achse an, da die Verteilung
in Boxplots normalerweise vertikal dargestellt wird.
plot_3 <- ggplot(kursdata_anon, aes(y = lezufr))
plot_3

Die Diagrammart geom_boxplot und die Farben werden
bestimmt. Mit coef=0 werden die Linien ober und
unterhalb des Boxplots eleminiert, die jeweils das das 1,5-Fache des IQR
messen und insbesondere zur kompakten Darstellung bei Daten mit vielen
Messpunkten sinnvoll sind.
plot_3 <- ggplot(kursdata_anon, aes(y = lezufr))+
geom_boxplot(colour = "blue", fill = "cyan", coef=0)
plot_3

Die “Range” der y-Achse wird mit ylim(0,100)
bestimmt. So kann die y-Achse erweitert werden, damit nicht nur der
empirisch realisierte Bereich abgebildet wird. In unserem Beispiel gibt
es Leute mit Lebenszufriedenheit (0) aber auch (100). Das heisst die
Range ist maximal. Es hätte jedoch auch sein können, dass niemand eine
Lebenszufriedenheit unter (30) angegeben hat. Dann würde die Skala auf
der y-Achse bei 30 enden. Mit ylim(0,100) könnten wir
sie trotzdem bis 0 ziehen und somit den ganzen theoretischen
Wertebereich der Variable lezufr anzeigen. Mit
labs() werden wie gewohnt Beschriftungen
vorgenommen.
plot_3 <- ggplot(kursdata_anon, aes(y = lezufr))+
geom_boxplot(colour = "blue", fill = "cyan", coef=0)+
ylim(0,100) +
labs(y = "Lebenszufriedeheitsskala",
title = "Verteilung der Lebenszufriedenheit",
subtitle = "0 = gar nicht zufrieden, 100 = sehr zufrieden",
caption = "Quelle: Kursbefragung Statistik I (n=...)")
plot_3

Die x-Achse ist im Falle einer univariaten Verteilung mit einem
Boxplot nicht von Bedeutung. Wir entfernen also die Beschriftung der
x-Achse über labs und die Skala über
aes.
plot_3 <- ggplot(kursdata_anon, aes(x = "",y = lezufr))+
geom_boxplot(colour = "blue", fill = "cyan", coef=0) +
ylim(0,100)+
labs(y = "Lebenszufriedeheitsskala", x = "",
title = "Verteilung der Lebenszufriedenheit",
subtitle = "0 = gar nicht zufrieden, 100 = sehr zufrieden",
caption = "Quelle: Kursbefragung Statistik I (n=...)")
plot_3

Schlussendlich wollen wir ein Rauten-Symbol für den Mittelwert/mean
einfügen. Dafür benutzen wir stat_summary, über welchen
wir Wert, Form und Farbe der Raute definieren können.
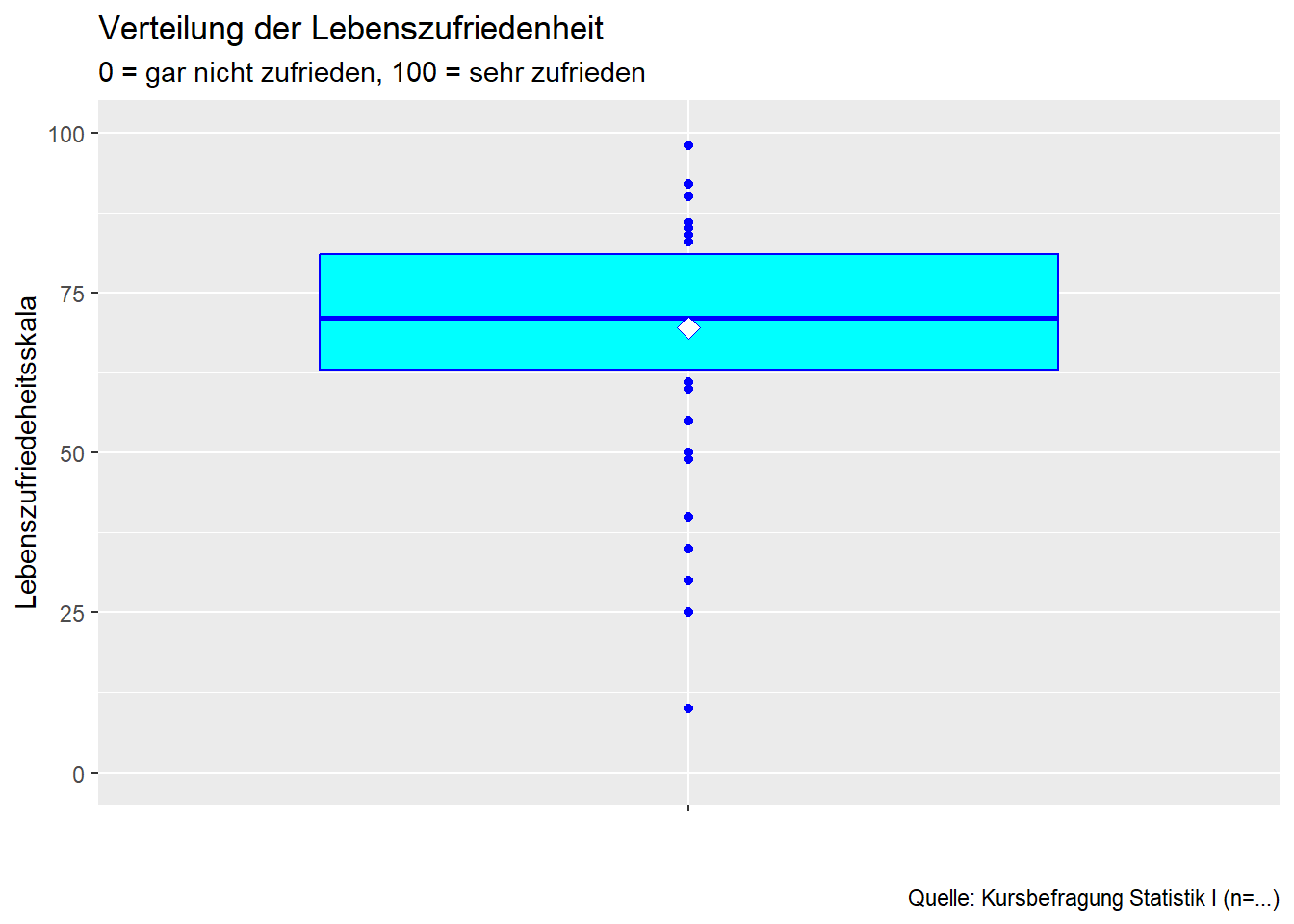
plot_3 <- ggplot(kursdata_anon, aes(x = "",y = lezufr))+
geom_boxplot(colour = "blue", fill = "cyan", coef=0) +
ylim(0,100)+
labs(y = "Lebenszufriedeheitsskala", x = "",
title = "Verteilung der Lebenszufriedenheit",
subtitle = "0 = gar nicht zufrieden, 100 = sehr zufrieden",
caption = "Quelle: Kursbefragung Statistik I (n=...)") +
stat_summary(fun=mean, geom="point", shape=23, size=3, color="blue", fill="white")
plot_3
3. Export von Grafiken
Möglichkeit 1: Die quick & dirty “in 15 Minuten beginnt der
Vortrag”-Variante
Du kannst die zuletzt aufgerufene Grafik im R-Studio Fenster unten
rechts (unter “Plots”) durch manuelles Verschieben von Fenstergrösse und
-verhältnis so kalibrieren, dass Seitenabmessung, Schriftgrösse etc.
stimmig sind. Nun kannst du den Graph per rechtsklick kopieren (oder mit
einem Bildschirmausschnitt, zweckmässig über ein Snippet Tool o.ä., die
Grafik ausschneiden) und in eine Präsentation oder sonstigen File
einfügen. Das Ergebnis ist für präsentationszwecke meist ok. Für die
Einbindung in schriftliche Dokumente, Poster etc ist diese Variante
aufgrund der mittelmässigen Qualität und fehlender Replizierbarkeit
gleichwohl nicht geeignet.
Möglichkeit 2: Skriptbasiert mit dem generischen
R-Grafiktreiber
Alternativ können die Grafiken über das Skript exportiert werden. Die
Grafik wird dann automatisch in dem als working directory
definierten Ordner abgespeichert.
png("boxplot.png", width = 12, height = 12, units = "cm", res = 300)
plot(plot_3)
dev.off()
pdf("boxplot.pdf")
plot(plot_3)
dev.off()
Auf diese Weise können verschiedene Grafikformate realisiert werden -
im Beispielcode oben wurde die Abbildung sowohl im .png-Format
als auch als pdf angelegt. Diese Formate sind je nach
Anwendungskontext und Grafikvorgaben mal mehr, mal weniger geeignet.
pdf() kreiert auflösungsunabhängige Vektorgrafiken, was
oft attraktiv ist, manchmal aber auch nicht funktioniert.
png() funktioniert dagegen fast immer, erzeugt aber
extrem grosse Dateien wenn die Auflösung z.B. für eine Postergrafik
hinreichend sein soll.
In keinem der gängigen Speicherformate bleibt es nach unserer
Erfahrung aus, sich schrittweise zur optimalen Kombination von
Exportkonfiguration und ggf. auch Schriftgrösseneinstellungen im
ggplot-Befehl vorzuarbeiten.
Möglichkeit 3: Skriptbasiert mit ggsave()
Letzter Punkt gilt grundsätzlich auch für die dritte Exportvariante.
Das Kommando ggsave() ist spezifisch auf
ggplot()-produzierte Grafiken ausgerichtet und lässt sich dann oft
komfortabler nutzen als die generischen R-Grafiktreiber. Möglicherweise
nutzt es häufig einen sinnvollen Grössen-, Auflösungs- und
Seitenverhältnisdefault beim Speichern von Grafiken ohne dezidierte
Spezifikation - darauf würde ich mich aber nicht verlassen und lieber
stets die Konfiguration selbst anpassen. Auch ggsave()
kann in verschiedene Formate exportieren.
ggsave(filename = "boxplot_ggsave1.png", plot=plot_3)
ggsave(filename = "boxplot_ggsave2.png", plot=plot_3, height=10, width=10, units = "cm", dpi = 600)
ggsave(filename = "boxplot_ggsave3.png", plot=plot_3, height=5, width=5, units = "cm", dpi = 600)
ggsave(filename = "boxplot_ggsave4.png", plot=plot_3, height=40, width=40, units = "cm", dpi = 600)
ggsave(filename = "boxplot_ggsave3.pdf", height=4, width=4)
plot_3 <- ggplot(kursdata_anon, aes(x = "", y = lezufr)) +
geom_boxplot(colour = "blue", fill = "cyan", coef = 0, outlier.size = 4) +
ylim(0, 100) +
labs(
y = "Lebenszufriedeheitsskala",
x = "",
title = "Verteilung der Lebenszufriedenheit",
subtitle = "0 = gar nicht zufrieden, 100 = sehr zufrieden",
caption = "Quelle: Kursbefragung Statistik I (n=...)"
) +
stat_summary(fun = mean, geom = "point", shape = 23, size = 10, color = "blue", fill = "white") +
theme(
plot.title = element_text(size = 58),
plot.subtitle = element_text(size = 32),
axis.title.y = element_text(size = 32),
axis.text.y = element_text(size = 32),
plot.caption = element_text(size = 32)
)
Folgende Texte empfehlen wir euch zur Lektüre und Vertiefung:
BCP Kapitel 4: Beckerman,
A.P., Childs, D.Z., Petchey, O.L. (2017): Getting Started with R.
Oxford: University Press.
GW Kapitel 3: Wickham, Hadley
und Garrett Grolemund (2018): R for Data Science. Import, Tidy,
Transform, Visualize, and Model Data.
SVS Kapitel 4.3: Marco R.
Steenbergen, Kushtrim Veseli, Benjamin Schlegel (2015): Working with
Descriptive and Inferential Statistics in R. Script.